Python: Tamaño de píxel y dimensiones#
Esta sección proporciona un poco de información sobre cómo trabajar con tamaños de píxel y dimensiones en Python… que es un poco más complicado de lo que uno podría esperar a primera vista.
# First, our usual default imports
import sys
sys.path.append('../../../')
from helpers import *
import matplotlib.pyplot as plt
import numpy as np
Tamaño de píxel#
Comprobar el tamaño de los píxeles en Python ha sido, en mi opinión, un poco engorroso, porque las bibliotecas habituales utilizadas para leer imágenes no siempre facilitan el acceso a esa información.
Sin embargo, la situación está mejorando.
A continuación, veremos cómo acceder a la información sobre el tamaño de los píxeles utilizando dos conocidas bibliotecas de lectura de imágenes:
imageio- de uso muy común, facilita la lectura de muchos tipos comunes de imágenes.AICSImageIO- que es un poco más complejo, pero tiene algunas características extremadamente útiles para trabajar con imágenes científicas.
ImageIO#
Para explorar el tamaño de los píxeles con imageio, volvamos a la imagen neuronal utilizada en el capítulo “Canales y colores”.
El siguiente código muestra cómo podemos leer tanto los valores de los píxeles como los metadatos.
# In preparation for the future, we'll use the 'v3' imageio process
import imageio.v3 as iio
# Get the path to the image (this is a specific helper function for this book)
path = find_image('Rat_Hippocampal_Neuron.tif')[0]
# Read the pixel values
im_iio = iio.imread(path)
# Read & print the metadata
metadata = iio.immeta(path)
print(metadata)
{'byteorder': '>', 'is_bif': False, 'is_micromanager': False, 'is_tiffep': False, 'is_fluoview': False, 'is_stk': False, 'is_ndpi': False, 'is_ome': False, 'is_mrc': False, 'is_multipage': False, 'is_nih': False, 'is_scanimage': False, 'is_shaped': False, 'is_streak': False, 'is_metaseries': False, 'is_fei': False, 'is_gdal': False, 'is_svs': False, 'is_uniform': False, 'is_tvips': False, 'is_mediacy': False, 'is_epics': False, 'is_astrotiff': False, 'is_eer': False, 'is_imagej': True, 'ImageJ': '1.44o', 'images': 5, 'channels': 5, 'mode': 'color', 'unit': 'um', 'loop': False, 'min': 472.0, 'max': 2436.0, 'Info': 'This is a composite confocal image of primary hippocampal neurons.\nHigh affinity bungarotoxin receptors were stained with fluorescent \nbungarotoxin (c=1). The nicotinic acetylcholine alpha7 subunit is \nimmunofluorescently labeled (c=2). A nAChR chaperone protein fused \nwith CFP was transiently transfected into the neurons (c=3). Nuclei \nwere dyed with Hoechst (c=4). A Nomarski optics image shows the \nmorphology of the neuron (c=5). Image is courtesy of John Alexander.\n', 'Ranges': (472.0, 2436.0, 548.3125, 2935.75, 504.84765625, 942.6484375, 518.359375, 3141.347237880496, 1937.9375, 3136.4940476190477), 'LUTs': [array([[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12,
13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25,
26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38,
39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51,
52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64,
65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77,
78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90,
91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 101, 102, 103,
104, 105, 106, 107, 108, 109, 110, 111, 112, 113, 114, 115, 116,
117, 118, 119, 120, 121, 122, 123, 124, 125, 126, 127, 128, 129,
130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142,
143, 144, 145, 146, 147, 148, 149, 150, 151, 152, 153, 154, 155,
156, 157, 158, 159, 160, 161, 162, 163, 164, 165, 166, 167, 168,
169, 170, 171, 172, 173, 174, 175, 176, 177, 178, 179, 180, 181,
182, 183, 184, 185, 186, 187, 188, 189, 190, 191, 192, 193, 194,
195, 196, 197, 198, 199, 200, 201, 202, 203, 204, 205, 206, 207,
208, 209, 210, 211, 212, 213, 214, 215, 216, 217, 218, 219, 220,
221, 222, 223, 224, 225, 226, 227, 228, 229, 230, 231, 232, 233,
234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244, 245, 246,
247, 248, 249, 250, 251, 252, 253, 254, 255],
[ 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0],
[ 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0]], dtype=uint8), array([[ 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0],
[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12,
13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25,
26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38,
39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51,
52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64,
65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77,
78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90,
91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 101, 102, 103,
104, 105, 106, 107, 108, 109, 110, 111, 112, 113, 114, 115, 116,
117, 118, 119, 120, 121, 122, 123, 124, 125, 126, 127, 128, 129,
130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142,
143, 144, 145, 146, 147, 148, 149, 150, 151, 152, 153, 154, 155,
156, 157, 158, 159, 160, 161, 162, 163, 164, 165, 166, 167, 168,
169, 170, 171, 172, 173, 174, 175, 176, 177, 178, 179, 180, 181,
182, 183, 184, 185, 186, 187, 188, 189, 190, 191, 192, 193, 194,
195, 196, 197, 198, 199, 200, 201, 202, 203, 204, 205, 206, 207,
208, 209, 210, 211, 212, 213, 214, 215, 216, 217, 218, 219, 220,
221, 222, 223, 224, 225, 226, 227, 228, 229, 230, 231, 232, 233,
234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244, 245, 246,
247, 248, 249, 250, 251, 252, 253, 254, 255],
[ 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0]], dtype=uint8), array([[ 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0],
[ 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0],
[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12,
13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25,
26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38,
39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51,
52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64,
65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77,
78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90,
91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 101, 102, 103,
104, 105, 106, 107, 108, 109, 110, 111, 112, 113, 114, 115, 116,
117, 118, 119, 120, 121, 122, 123, 124, 125, 126, 127, 128, 129,
130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142,
143, 144, 145, 146, 147, 148, 149, 150, 151, 152, 153, 154, 155,
156, 157, 158, 159, 160, 161, 162, 163, 164, 165, 166, 167, 168,
169, 170, 171, 172, 173, 174, 175, 176, 177, 178, 179, 180, 181,
182, 183, 184, 185, 186, 187, 188, 189, 190, 191, 192, 193, 194,
195, 196, 197, 198, 199, 200, 201, 202, 203, 204, 205, 206, 207,
208, 209, 210, 211, 212, 213, 214, 215, 216, 217, 218, 219, 220,
221, 222, 223, 224, 225, 226, 227, 228, 229, 230, 231, 232, 233,
234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244, 245, 246,
247, 248, 249, 250, 251, 252, 253, 254, 255]], dtype=uint8), array([[ 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0],
[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12,
13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25,
26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38,
39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51,
52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64,
65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77,
78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90,
91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 101, 102, 103,
104, 105, 106, 107, 108, 109, 110, 111, 112, 113, 114, 115, 116,
117, 118, 119, 120, 121, 122, 123, 124, 125, 126, 127, 128, 129,
130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142,
143, 144, 145, 146, 147, 148, 149, 150, 151, 152, 153, 154, 155,
156, 157, 158, 159, 160, 161, 162, 163, 164, 165, 166, 167, 168,
169, 170, 171, 172, 173, 174, 175, 176, 177, 178, 179, 180, 181,
182, 183, 184, 185, 186, 187, 188, 189, 190, 191, 192, 193, 194,
195, 196, 197, 198, 199, 200, 201, 202, 203, 204, 205, 206, 207,
208, 209, 210, 211, 212, 213, 214, 215, 216, 217, 218, 219, 220,
221, 222, 223, 224, 225, 226, 227, 228, 229, 230, 231, 232, 233,
234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244, 245, 246,
247, 248, 249, 250, 251, 252, 253, 254, 255],
[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12,
13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25,
26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38,
39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51,
52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64,
65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77,
78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90,
91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 101, 102, 103,
104, 105, 106, 107, 108, 109, 110, 111, 112, 113, 114, 115, 116,
117, 118, 119, 120, 121, 122, 123, 124, 125, 126, 127, 128, 129,
130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142,
143, 144, 145, 146, 147, 148, 149, 150, 151, 152, 153, 154, 155,
156, 157, 158, 159, 160, 161, 162, 163, 164, 165, 166, 167, 168,
169, 170, 171, 172, 173, 174, 175, 176, 177, 178, 179, 180, 181,
182, 183, 184, 185, 186, 187, 188, 189, 190, 191, 192, 193, 194,
195, 196, 197, 198, 199, 200, 201, 202, 203, 204, 205, 206, 207,
208, 209, 210, 211, 212, 213, 214, 215, 216, 217, 218, 219, 220,
221, 222, 223, 224, 225, 226, 227, 228, 229, 230, 231, 232, 233,
234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244, 245, 246,
247, 248, 249, 250, 251, 252, 253, 254, 255]], dtype=uint8), array([[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12,
13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25,
26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38,
39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51,
52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64,
65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77,
78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90,
91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 101, 102, 103,
104, 105, 106, 107, 108, 109, 110, 111, 112, 113, 114, 115, 116,
117, 118, 119, 120, 121, 122, 123, 124, 125, 126, 127, 128, 129,
130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142,
143, 144, 145, 146, 147, 148, 149, 150, 151, 152, 153, 154, 155,
156, 157, 158, 159, 160, 161, 162, 163, 164, 165, 166, 167, 168,
169, 170, 171, 172, 173, 174, 175, 176, 177, 178, 179, 180, 181,
182, 183, 184, 185, 186, 187, 188, 189, 190, 191, 192, 193, 194,
195, 196, 197, 198, 199, 200, 201, 202, 203, 204, 205, 206, 207,
208, 209, 210, 211, 212, 213, 214, 215, 216, 217, 218, 219, 220,
221, 222, 223, 224, 225, 226, 227, 228, 229, 230, 231, 232, 233,
234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244, 245, 246,
247, 248, 249, 250, 251, 252, 253, 254, 255],
[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12,
13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25,
26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38,
39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51,
52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64,
65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77,
78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90,
91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 101, 102, 103,
104, 105, 106, 107, 108, 109, 110, 111, 112, 113, 114, 115, 116,
117, 118, 119, 120, 121, 122, 123, 124, 125, 126, 127, 128, 129,
130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142,
143, 144, 145, 146, 147, 148, 149, 150, 151, 152, 153, 154, 155,
156, 157, 158, 159, 160, 161, 162, 163, 164, 165, 166, 167, 168,
169, 170, 171, 172, 173, 174, 175, 176, 177, 178, 179, 180, 181,
182, 183, 184, 185, 186, 187, 188, 189, 190, 191, 192, 193, 194,
195, 196, 197, 198, 199, 200, 201, 202, 203, 204, 205, 206, 207,
208, 209, 210, 211, 212, 213, 214, 215, 216, 217, 218, 219, 220,
221, 222, 223, 224, 225, 226, 227, 228, 229, 230, 231, 232, 233,
234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244, 245, 246,
247, 248, 249, 250, 251, 252, 253, 254, 255],
[ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12,
13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25,
26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38,
39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51,
52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64,
65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77,
78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90,
91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 101, 102, 103,
104, 105, 106, 107, 108, 109, 110, 111, 112, 113, 114, 115, 116,
117, 118, 119, 120, 121, 122, 123, 124, 125, 126, 127, 128, 129,
130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142,
143, 144, 145, 146, 147, 148, 149, 150, 151, 152, 153, 154, 155,
156, 157, 158, 159, 160, 161, 162, 163, 164, 165, 166, 167, 168,
169, 170, 171, 172, 173, 174, 175, 176, 177, 178, 179, 180, 181,
182, 183, 184, 185, 186, 187, 188, 189, 190, 191, 192, 193, 194,
195, 196, 197, 198, 199, 200, 201, 202, 203, 204, 205, 206, 207,
208, 209, 210, 211, 212, 213, 214, 215, 216, 217, 218, 219, 220,
221, 222, 223, 224, 225, 226, 227, 228, 229, 230, 231, 232, 233,
234, 235, 236, 237, 238, 239, 240, 241, 242, 243, 244, 245, 246,
247, 248, 249, 250, 251, 252, 253, 254, 255]], dtype=uint8)], 'is_philips': False, 'is_qpi': False, 'is_scn': False, 'is_andor': False, 'is_volumetric': False, 'is_lsm': False, 'is_geotiff': False, 'is_sem': False, 'is_subifd': False, 'is_vista': False, 'is_pilatus': False, 'is_dng': False, 'is_ndtiff': False, 'is_mdgel': False, 'is_sis': False}
Los metadatos contienen mucha información, incluso tablas de consulta.
Imprimiéndolo un poco más bonito (y saltándonos las LUTs), obtenemos:
for k, v in metadata.items():
if not 'LUTs' in k:
print(f'{k}: {v}')
byteorder: >
is_bif: False
is_micromanager: False
is_tiffep: False
is_fluoview: False
is_stk: False
is_ndpi: False
is_ome: False
is_mrc: False
is_multipage: False
is_nih: False
is_scanimage: False
is_shaped: False
is_streak: False
is_metaseries: False
is_fei: False
is_gdal: False
is_svs: False
is_uniform: False
is_tvips: False
is_mediacy: False
is_epics: False
is_astrotiff: False
is_eer: False
is_imagej: True
ImageJ: 1.44o
images: 5
channels: 5
mode: color
unit: um
loop: False
min: 472.0
max: 2436.0
Info: This is a composite confocal image of primary hippocampal neurons.
High affinity bungarotoxin receptors were stained with fluorescent
bungarotoxin (c=1). The nicotinic acetylcholine alpha7 subunit is
immunofluorescently labeled (c=2). A nAChR chaperone protein fused
with CFP was transiently transfected into the neurons (c=3). Nuclei
were dyed with Hoechst (c=4). A Nomarski optics image shows the
morphology of the neuron (c=5). Image is courtesy of John Alexander.
Ranges: (472.0, 2436.0, 548.3125, 2935.75, 504.84765625, 942.6484375, 518.359375, 3141.347237880496, 1937.9375, 3136.4940476190477)
is_philips: False
is_qpi: False
is_scn: False
is_andor: False
is_volumetric: False
is_lsm: False
is_geotiff: False
is_sem: False
is_subifd: False
is_vista: False
is_pilatus: False
is_dng: False
is_ndtiff: False
is_mdgel: False
is_sis: False
En realidad, estos metadatos son bastante específicos de ImageJ, y otros TIFF pueden proporcionar metadatos muy diferentes.
Podemos ver la versión de ImageJ que escribió el archivo, pero elegir el elemento clave que queremos aquí - el tamaño de píxel - no es tan fácil.
Ver «unit=um» es alentador, pero no suficiente.
Podemos explorar un poco más con “propiedades”, que imageio describe como “un conjunto curado de metadatos estandarizados”.
properties = iio.improps(path, extension=".tif")
print(properties)
ImageProperties(shape=(512, 512), dtype=dtype('uint16'), n_images=None, is_batch=False, spacing=(6.25, 6.25))
Aquí, el spacing=(6.25, 6.25) parece prometedor.
Es tentador suponer que eso significa que la anchura y la altura del píxel son ambas de 6.25 µm - sin embargo si compruebo la misma imagen en el propio ImageJ, veo que la anchura y la altura del píxel son en realidad de 0.16 µm… lo que resulta ser igual a 1.0/6.25 µm.
print(1.0 / 6.25)
0.16
Por lo tanto, la información está en los metadatos, pero es muy fácil malinterpretarla, y ni siquiera está garantizado que sea correcta si la imagen fue escrita por algún otro software.
Así que mientras imageio es excelente para leer imágenes fácilmente - generalmente sólo un rápido im = imread(path) - no es necesariamente lo mejor para usar cuando el tamaño de los píxeles (u otros metadatos) importan.
Uso de AICSImageIO#
La mejor alternativa que conozco para trabajar con imágenes científicas (especialmente biomédicas) es AICSImageIO. Se trata de una biblioteca de Python realmente útil que estandariza la lectura y escritura de múltiples formatos de archivo - y, dependiendo de cómo esté instalada, puede incluso acceder a muchos más formatos raros de archivo con la ayuda de Bio-Formats.
Aunque es posible utilizar una versión de imread con AICSImageIO, vale la pena aprender la forma alternativa de hacer las cosas creando un objeto AICSImage. Esto nos proporciona una forma de acceder a los píxeles y a muchas otras cosas útiles siempre que las necesitemos.
from aicsimageio.aics_image import AICSImage
# Create an AICSImage
img_aics = AICSImage(path)
# Print its main attributes
print(img_aics)
for d in dir(img_aics):
if not d.startswith('_'):
print(d)
<AICSImage [Reader: TiffReader, Image-is-in-Memory: False]>
ReaderClass
SUPPORTED_READERS
channel_names
current_scene
current_scene_index
dask_data
data
determine_reader
dims
dtype
get_dask_stack
get_image_dask_data
get_image_data
get_mosaic_tile_position
get_stack
get_xarray_dask_stack
get_xarray_stack
metadata
mosaic_tile_dims
ome_metadata
physical_pixel_sizes
reader
reader_path
reader_paths
save
scenes
set_scene
shape
xarray_dask_data
xarray_data
/usr/share/miniconda/envs/bioimage-book/lib/python3.10/site-packages/pydantic/_migration.py:283: UserWarning: `pydantic.error_wrappers:ValidationError` has been moved to `pydantic:ValidationError`.
warnings.warn(f'`{import_path}` has been moved to `{new_location}`.')
A partir de aquí, podemos ver inmediatamente el atributo que nos proporcionará directamente el tamaño de los píxeles.
print(img_aics.physical_pixel_sizes)
PhysicalPixelSizes(Z=None, Y=0.16, X=0.16)
Una cosa quizás no obvia que hay que saber al usar AICSImageIO es que AICSImage no es un array NumPy normal del tipo que devolvería imageio.imread. Más bien, si quieres eso, tienes que solicitar los datos.
A partir de este conocimiento, podemos comprobar que tenemos el mismo valor medio de píxel para ambos, como una forma rápida de asegurarnos de que los valores reales de píxel probablemente coincidan.
print(f'Mean pixel value from imageio: {im_iio.mean():.2f} (total pixel count {im_iio.size})')
print(f'Mean pixel value from AICSImageIO: {img_aics.data.mean():.2f} (total pixel count {img_aics.data.size})')
Mean pixel value from imageio: 1038.69 (total pixel count 1310720)
Mean pixel value from AICSImageIO: 1038.69 (total pixel count 1310720)
Dimensiones#
Formas y dimensiones de las matrices#
Hemos visto cómo dos librerías diferentes pueden permitirnos leer los mismos valores de píxeles como matrices NumPy y extraer información sobre el tamaño de los píxeles.
Podríamos esperar que las matrices NumPy que representan los valores de los píxeles fueran iguales, pero en realidad no podemos contar con ello.
NumPy es increíblemente flexible cuando se trata de manejar matrices multidimensionales. Y aunque esa flexibilidad puede ser realmente útil, también puede complicar las cosas.
Para verlo en acción, comprobemos las dimensiones de las imágenes que leemos utilizando imageio y AICSImageIO.
# Print shape of image read by imageio
import imageio.v3 as iio
path = find_image('Rat_Hippocampal_Neuron.tif')[0]
im_iio = iio.imread(path)
print(f'Shape of image read by imageio: {im_iio.shape}')
# Print shape of image read by AICSImageIO
from aicsimageio.aics_image import AICSImage
im_aics = AICSImage(path).data
print(f'Shape of image read by AICSImageIO: {im_aics.shape}')
print(f'Arrays the same? {np.array_equal(im_aics, im_iio)}')
Shape of image read by imageio: (5, 512, 512)
Shape of image read by AICSImageIO: (1, 5, 1, 512, 512)
Arrays the same? False
Podemos ver que el número de píxeles es el mismo, pero hay algunas dimensiones “unicas” adicionales metidas en los resultados de AICSImageIO (es decir, con longitud 1).
Afortunadamente, podemos eliminarlos fácilmente con un np.squeeze - y acabar con las mismas matrices.
im_aics_squeezed = np.squeeze(im_aics)
print(f'Shape of image read by AICSImageIO & squeezed: {im_aics_squeezed.shape}')
print(f'Arrays the same? {np.array_equal(im_aics_squeezed, im_iio)}')
Shape of image read by AICSImageIO & squeezed: (5, 512, 512)
Arrays the same? True
Así que una pregunta natural es: **¿Por qué AICSImageIO ha introducido algunas dimensiones extra?
Antes de responder a eso, deberíamos preguntarnos otra cosa. **¿Qué tenemos exactamente a lo largo de la dimensión «5»?
Y aquí es donde las cosas no están muy claras con Imageio.
5 podría ser la anchura de la imagen, la altura de la imagen, el número de canales, el número de secciones z o el número de puntos temporales.
Basándonos en nuestro conocimiento de las imágenes y de las otras dimensiones, podemos suponer que el «5» no corresponde a la anchura ni a la altura de la imagen (el «512» parece más probable), por lo que probablemente sea una de las otras.
Pero el problema es que no tenemos forma de saberlo sin más información. Si no tenemos una fuente externa que nos lo diga, tenemos que hurgar en los metadatos o mirar el contenido para averiguar la respuesta.
# Loop through the first dimension and show images for each plane
n_slices = im_iio.shape[0]
plt.figure(figsize=(12, 4))
for ii in range(n_slices):
plt.subplot(1, n_slices, ii+1)
plt.title(f'Plane {ii+1}')
show_image(im_iio[ii], clip_percentile=1)
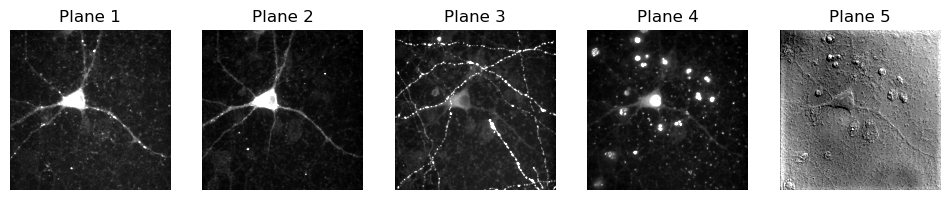
A mí me parece que tenemos 5 canales diferentes. Estoy haciendo algunas suposiciones… pero parecen suposiciones bastante seguras.
Sin embargo, AICSImageIO elimina esta ambigüedad de un par de maneras.
Puedes esperar que
AICSImagedevuelva un array 5D, con las dimensiones en un orden consistente: TCZYX` (aunque hay al menos una advertencia en la siguiente sección).Puede consultar fácilmente las dimensiones y el orden para estar seguro
image = AICSImage(path)
print(image.dims)
print(f'Shape: {image.dims.shape}')
print(f'Order: {image.dims.order}')
<Dimensions [T: 1, C: 5, Z: 1, Y: 512, X: 512]>
Shape: (1, 5, 1, 512, 512)
Order: TCZYX
¿Dónde están mis canales?#
Hemos visto anteriormente que imageio puede devolver una imagen de 5 canales con los canales en primer lugar. Nuestra pregunta aquí es: ¿lo hace siempre?
La respuesta, por desgracia, es no. La ubicación de las dimensiones de los canales es dolorosamente incierta en Python, y a menudo diferentes herramientas esperan que esté en diferentes lugares.
O a veces la misma herramienta puede ponerlo en un lugar diferente.
Para verlo en acción, vamos a leer una simple imagen RGB con imageio.
path = find_image('leaf.jpg')[0]
im_iio = iio.imread(path)
print(im_iio.shape)
(446, 507, 3)
Una imagen RGB tiene 3 canales - rojo, verde y azul - pero parece que de repente tenemos la dimensión de los canales en último lugar.
¿Por qué?
No tengo una explicación muy satisfactoria, excepto decir que para RGB eso es a menudo lo que quieres porque matplotlib espera que los canales sean los últimos, y a menudo usamos matplotlib para mostrar imágenes.
# Show an RGB image with channels-last using matplotlib
from matplotlib import pyplot as plt
path = find_image('leaf.jpg')[0]
im_iio = iio.imread(path)
plt.imshow(im_iio)
plt.show()
Sin embargo, no es siempre lo que quieres, y si consigues un aprendizaje profundo suficiente, te encontrarás a menudo con la pregunta «canales primero» o «canales después».
Con esto en mente, podemos usar NumPy para cambiar de los llamados “canales-últimos” a “canales-primeros” - pero a matplotlib no le gustará mucho.
# *Try* to show an RGB image with channels-first using matplotlib
im_iio_channels_first = np.moveaxis(im_iio, source=-1, destination=0)
print(f'My new shape: {im_iio_channels_first.shape}')
try:
plt.imshow(im_iio_channels_first)
plt.show()
except Exception as ex:
print(f"I can't show that, sorry! {ex}")
My new shape: (3, 446, 507)
I can't show that, sorry! Invalid shape (3, 446, 507) for image data
Así que imageio puede obtener canales al principio o al final. Para RGB, parece preferir el final.
¿Qué hace AICSImageIO?
Ya que dije que AICSImageIO es consistente, me gustaría decir que pone los canales en el mismo lugar para la imagen RGB y la de 5 canales… pero no. También trata RGB como un caso especial.
image = AICSImage(path)
print(image.shape)
print(image.dims.order)
(1, 1, 1, 446, 507, 3)
TCZYXS
Es un poco difícil de encontrar, pero la documentación de AICSImageIO menciona que se pueden esperar 5 dimensiones para imágenes no RGB, pero las imágenes RGB tienen 6 dimensiones - donde la sexta se llama S para Samples.
Lo bueno es que, suponiendo que no tienes nada más en marcha con las 3 primeras dimensiones - es decir, son sólo (1, 1, 1) - un simple np.squeeze es suficiente para convertir la matriz de píxeles en un formato RGB de último canal, que sea amigable con matplotlib.
Más dimensiones#
Terminaremos esta sección analizando una imagen con 2 canales y 25 cortes z.
Ya que ahora sabes cómo explorar las dimensiones a detalle, usaré mi función de ayuda
load_imagepor conveniencia… que devuelve un array NumPy que está pre-exprimido para eliminar cualquier dimensión única (singleton).
im = load_image('confocal-series.zip')
print(f'Shape: {im.shape}')
Shape: (25, 2, 400, 400)
Como ya hemos visto cómo visualizar imágenes multicanal en el último capítulo, vamos a extraer aquí un solo canal.
# Channels come second
# This gives us all the z-slices (:), the first channel (0), everything else (...)
im_single = im[:, 0, ...]
print(f'New shape: {im_single.shape}')
New shape: (25, 400, 400)
En este punto, NumPy se vuelve bastante divertido - porque es tan fácil hacer cosas a lo largo de diferentes dimensiones.
Por ejemplo, podemos generar rápidamente diferentes proyecciones z.
plt.figure(figsize=(16, 4))
plt.subplot(1, 4, 1)
plt.imshow(im_single.max(axis=0))
plt.axis(False)
plt.title('Max z-projection')
plt.subplot(1, 4, 2)
plt.imshow(im_single.min(axis=0))
plt.axis(False)
plt.title('Min z-projection')
plt.subplot(1, 4, 3)
plt.imshow(im_single.mean(axis=0))
plt.axis(False)
plt.title('Mean z-projection')
plt.subplot(1, 4, 4)
plt.imshow(im_single.std(axis=0))
plt.axis(False)
plt.title('Std.dev. z-projection')
plt.show()
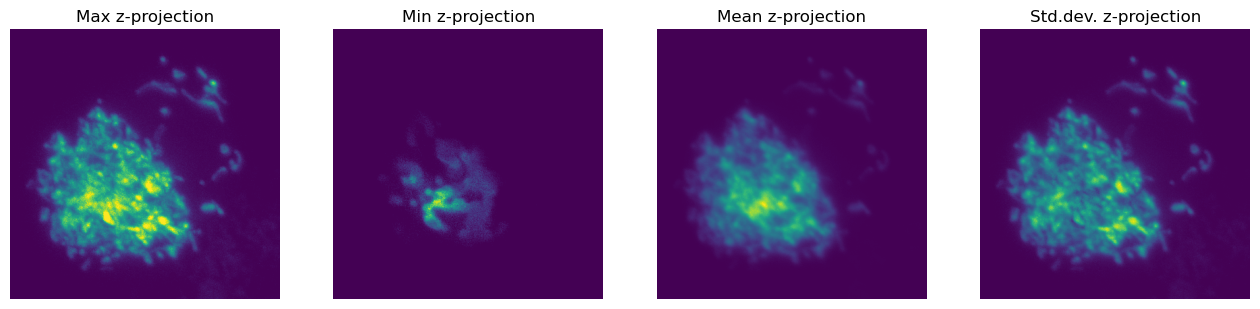
Pero no estamos limitados a proyectar a lo largo de z - podemos simplemente cambiar el valor del eje y proyectar a lo largo de alguna otra dimensión.
Ten en cuenta que esto no hará ninguna corrección para las diferencias en el tamaño de píxel en xy vs z. Con sólo 25 secciones z, estas proyecciones se ven muy aplastadas.
plt.figure(figsize=(16, 4))
plt.subplot(1, 4, 1)
plt.imshow(im_single.max(axis=1))
plt.axis(False)
plt.title('Max y-projection')
plt.subplot(1, 4, 2)
plt.imshow(im_single.min(axis=1))
plt.axis(False)
plt.title('Min y-projection')
plt.subplot(1, 4, 3)
plt.imshow(im_single.mean(axis=1))
plt.axis(False)
plt.title('Mean y-projection')
plt.subplot(1, 4, 4)
plt.imshow(im_single.std(axis=1))
plt.axis(False)
plt.title('Std.dev. y-projection')
plt.show()

Y también podemos cortar donde queramos para obtener vistas ortogonales.
plt.figure(figsize=(8, 8))
plt.subplot(2, 2, 1)
plt.imshow(im_single[im_single.shape[0]//2, ...])
plt.axis(False)
plt.title('Middle z-slice')
plt.subplot(2, 2, 3)
plt.imshow(im_single[:, im_single.shape[1]//2, ...])
plt.axis(False)
plt.title('Middle row')
plt.subplot(2, 2, 2)
plt.imshow(im_single[..., im_single.shape[2]//2].transpose())
plt.axis(False)
plt.title('Middle column')
plt.tight_layout()
plt.show()


